钍基核裂变能全国重点实验室 |
|||
|
-钍基核能物理中心-
|
|||
熔盐机械工程技术部 |
|||
仪控工程技术部 |
|||
熔盐化学工程技术部 |
|||
|
-核能综合利用研究中心-
|
|||
材料研究部 |
|||
钍铀循环化学部 |
|||
应用化学技术部 |
|||
氚科学与工程技术部 |
|||
核与辐射安全技术部 |
|||
应用加速器技术部 |
|||
反应堆运行技术部 |
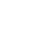



近日,中科院上海应用物理研究所研究人员实现了对界面酶分子的单分子实时荧光成像,通过对运动轨迹的分析发现酶分子的趋向运动(Chemotaxis)是平动与转动的竞争平衡结果, 相关工作发表于J. Am. Chem. Soc. 2017, 139, 17525−17532。
液体中的分子通常作无规则的布朗运动。而对于有催化活性的酶分子而言,它们可利用酶促反应过程中底物转化时释放的能量驱动其自身的运动。之前研究表明,酶分子运动的扩散系数与底物浓度呈正相关, 该现象被称为酶促分子马达。然而,酶分子是否存在类似细菌的趋向运动, 即酶分子是否主动向底物浓度高的方向扩散,是一个长期存疑的问题。例如,加州大学伯克利分校的Bustamante教授提出可以用“化学声效应 (chemoacoustic effect)”来解释这种酶促分子马达(Nature, 2015, 517, 227)。同期发表的对该文的评述中,美国宾州大学的Joshua Wand教授则指出,“看起来有可能酶分子进化出了寻找底物的能力,但此观点尚应存疑。”(It seems reasonable to think that enzymes might have evolved the capacity to seek ‘greener pastures’ of substrates. But this view is more complicated than it might at first look)
上海应物所物理生物学研究室博士研究生孙乐乐,高延静在研究员李迪、樊春海等指导下,基于DNA折纸术(DNA origami)发展了一种对界面上酶级联反应中的单个酶分子运动轨迹实时成像的方法,并对酶分子趋向运动机制作出了解释。他们以胆固醇修饰的DNA分子为锚定链,并利用胆固醇分子可以嵌入磷脂双层膜的特点,将酶分子固定在二维磷脂分子界面。研究发现,不同锚定手段可以控制酶分子在二维磷脂界面上的运动速度。其中,利用双链DNA动态锚定(dynamic tethering)的酶分子,可以在二维界面上自由扩散;而利用DNA折纸筏(origami raft)静态锚定(static tethering)的酶分子,则固定在磷脂膜上不动。基于此,他们在磷脂双分子层的二维界面上构建了一个酶级联反应:其中上游的葡萄糖氧化酶在界面上固定不动,而下游的过氧化氢酶可以在界面上自由扩散。利用全内反射显微镜,他们实时观测了单个酶分子的运动轨迹。研究发现:以葡萄糖氧化酶分子为坐标中心,下游的过氧化氢酶分子的平动速率确实随上游底物葡萄糖的增加而加快,但上下游酶分子的相对空间距离并未随酶促反应而改变。他们利用Einstein relation计算了界面上过氧化氢酶分子的自旋弛豫时间,发现过氧化氢酶的转动速率大大快于其平动速率,过氧化氢酶分子转动一圈的时间内,其平动距离仅为其自身直径的1/6, 因而,即使在酶促反应中,酶分子也并不存在趋向运动,趋向运动是平动与转动的竞争平衡结。
这一工作解释了酶促马达并未造成趋向运动的原因。该工作构建的控制界面上不同蛋白质分子运动速度的方法及对蛋白质运动轨迹实时成像的平台,可用于研究信号通路中多种蛋白质作用及抑制剂筛选,为研究蛋白质间的动态相互作用提供了新的工具。 (物理生物学研究室 供稿)